Se sabe que el síndrome respiratorio agudo severo, conocido como enfermedad COVID-19, causado por el virus SARS-CoV-2, se propaga a través de gotitas respiratorias y contactos cercanos.[1] La carga de COVID-19 fue extremadamente grave en Lombardía y el Valle del Po (norte de Italia),[2] una zona caracterizada por altas concentraciones de material particulado, que ya se sabe que produce efectos negativos en la salud humana.[3] Las cifras regionales disponibles para Italia al 12 de abril muestran que alrededor del 30% de las personas actualmente positivas aún viven en Lombardía (alrededor del 40% si se considera el total de casos confirmados desde el comienzo de la epidemia), seguida de Emilia Romaña (13,5%), Piamonte (10,5%) y Véneto (10%).[2] Estas cuatro regiones del Valle del Po representan el 80% del total de muertes registradas en Italia y el 65% de los ingresos en Unidades de Cuidados Intensivos.[2]
Una investigación realizada por la Escuela de Salud Pública de Harvard parece confirmar una asociación entre el aumento de las concentraciones de PM y las tasas de mortalidad por COVID-19 en los EE. UU. [4] En comunicaciones anteriores, hemos planteado la hipótesis de la posibilidad de que el virus SARS-CoV-2 pudiera estar presente en materia particulada (PM) durante la propagación de la infección, [5,6] en consonancia con la evidencia ya
disponible para otros virus.[7-15] Sin embargo, la cuestión del microbioma asociado a PM en el aire, especialmente en entornos urbanos, sigue siendo en gran medida poco investigada,[16] y, hasta la fecha, nadie ha llevado a cabo estudios experimentales específicamente dirigidos a confirmar o excluir la presencia del SARS-CoV-2 en PM.
Aquí presentamos los primeros resultados de los análisis que hemos realizado en 34 muestras de PM10 en el aire/exterior de un sitio industrial de la provincia de Bérgamo, recogidas con dos muestreadores de aire diferentes durante un período continuo de 3 semanas, del 21 de febrero al 13 de marzo.
Siguiendo la metodología descrita por Pan et al. en 2019 (para la recolección, el dimensionamiento de partículas y la detección de virus aerotransportados),[17] se recolectaron muestras de PM en filtros de fibra de cuarzo mediante un muestreador de aire gravimétrico de bajo volumen (38,3 l/min durante 23 h), de acuerdo con el método de referencia EN12341:2014 para el monitoreo de PM10. Las partículas quedaron atrapadas en los filtros con un 99,9 % de pureza típica.Retención de aerosoles, debidamente almacenada y entregada al laboratorio de Genómica Aplicada y Comparativa de la Universidad de Trieste. Dada la naturaleza ambiental de la muestra, presumiblemente rica en inhibidores de las ADN polimerasas, se procedió a la extracción de ARN utilizando el kit Quick RNA para microbios de suelo fecal, adaptado al tipo de filtro.[18] Se enrolló la mitad del filtro, con la parte superior hacia adentro.En un tubo de polipropileno de 5 ml, junto con las microesferas incluidas en el kit. A partir de 1 ml inicial de tampón de lisis, se obtuvieron aproximadamente 400 µl de solución, que se procesaron según los protocolos estándar, obteniendo un eluido final de 15 µl. Posteriormente, se utilizaron 5 µl para la prueba de SARS-CoV-2. Dado el origen particular de la muestra, se utilizó el qScript XLT 1-Step RT-qPCR ToughMix.[19] Los sistemas de amplificación fueron los del protocolo desarrollado por Corman et al., publicado en el sitio web de la OMS [20].
La prueba tenía como objetivo específico confirmar o descartar la presencia del ARN del SARS-CoV-2 en partículas. El primer análisis utilizó el gen E como marcador molecular y arrojó un impresionante resultado positivo en 15 de los 16 filtros, incluso si, como cabía esperar, el Ct se encontraba entre 36 y 38 ciclos.
Posteriormente, hemos replicado el análisis en 6 de los filtros positivos (ya positivos al “gen E”) utilizando como marcador molecular el “gen RtDR”, altamente específico para el SARS-CoV-2, alcanzando 5 resultados significativos de positividad; también se realizaron con éxito pruebas de control para excluir falsas positividades (Fig. 1).
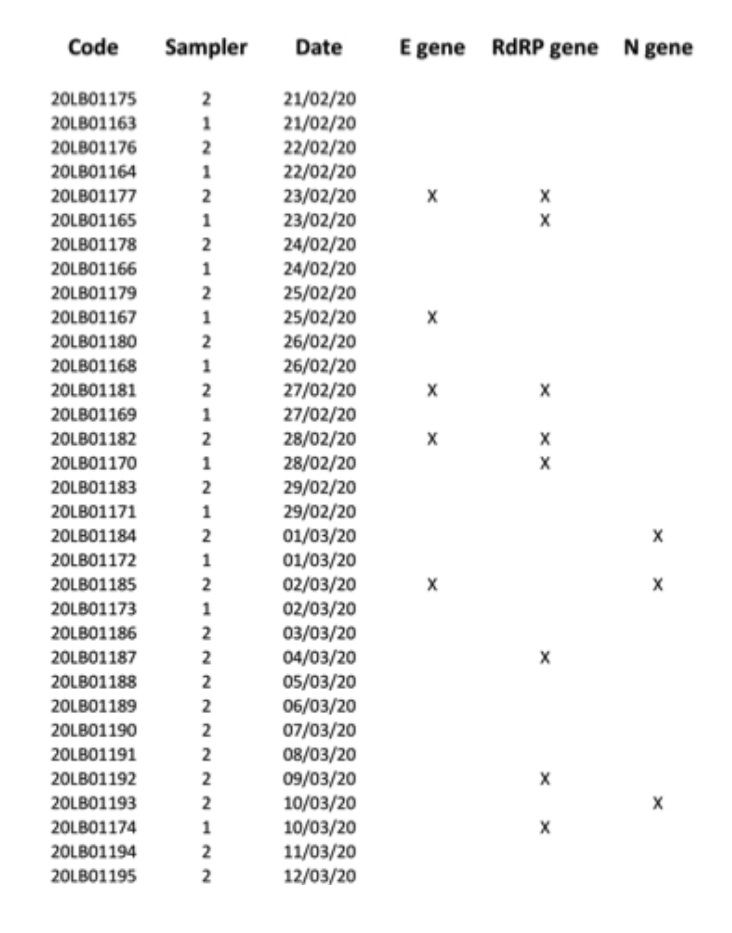
Para evitar que se agotara el escaso material de muestreo disponible, los ARN extraídos restantes se entregaron al Hospital Universitario local (uno de los centros clínicos autorizados por el Gobierno italiano para las pruebas de diagnóstico del SARS-CoV-2), con el fin de realizar una segunda prueba paralela a ciegas. Este segundo laboratorio clínico analizó 34 extracciones de ARN para los genes E, N y RdRP, informando 7 resultados positivos para al menos uno de los tres genes marcadores, con positividad confirmada por separado para los tres marcadores (Fig. 2). Debido a la naturaleza de la muestra, y considerando que el muestreo no se ha realizado con fines de diagnóstico clínico, sino para pruebas de contaminación ambiental (teniendo también en cuenta que los filtros se almacenaron durante al menos cuatro semanas antes de someterse a análisis genéticos moleculares, comoDebido al confinamiento en Italia, podemos confirmar que hemos demostrado razonablemente la presencia de ARN viral del SARS-CoV-2 mediante la detección del gen RtDR altamente específico en ocho filtros. Sin embargo, debido a la falta de materiales adicionales de los filtros, no pudimos repetir suficientes pruebas para obtener resultados positivos para los tres marcadores moleculares simultáneamente.
Esta es la primera evidencia preliminar de que el ARN del SARS-CoV-2 puede estar presente en partículas en suspensión en el exterior, lo que sugiere que, en condiciones de estabilidad atmosférica y altas concentraciones de PM, el SARS-CoV-2 podría crear cúmulos con PM en el exterior y, al reducir su coeficiente de difusión, aumentar la persistencia del virus en la atmósfera. Confirmaciones adicionales de este estudio preliminarSe están realizando estudios de evidencia que deberían incluir una evaluación en tiempo real de la vitalidad del SARS-CoV-2, así como de su virulencia al adsorberse en material particulado. Actualmente, no se pueden hacer suposiciones sobre la correlación entre la presencia del virus en PM y la progresión del brote de COVID-19. Otras cuestiones que deben abordarse específicamente son las concentraciones promedio de PM eventualmente.requerido para un potencial “efecto boost” del contagio (en caso de que se confirme que las PM podrían actuar como “portadoras” de los núcleos virales en gotitas), o incluso la posibilidad teórica de inmunización como consecuencia de exposiciones a dosis mínimas en umbrales inferiores de PM.
Fig.1 Curvas de amplificación de los genes E (A) y RdRP (B): las líneas verdes representan los filtros probados; las líneas cruzadasrepresenta extracciones de filtros de referencia; las líneas rojas representan la amplificación de las muestras positivas.
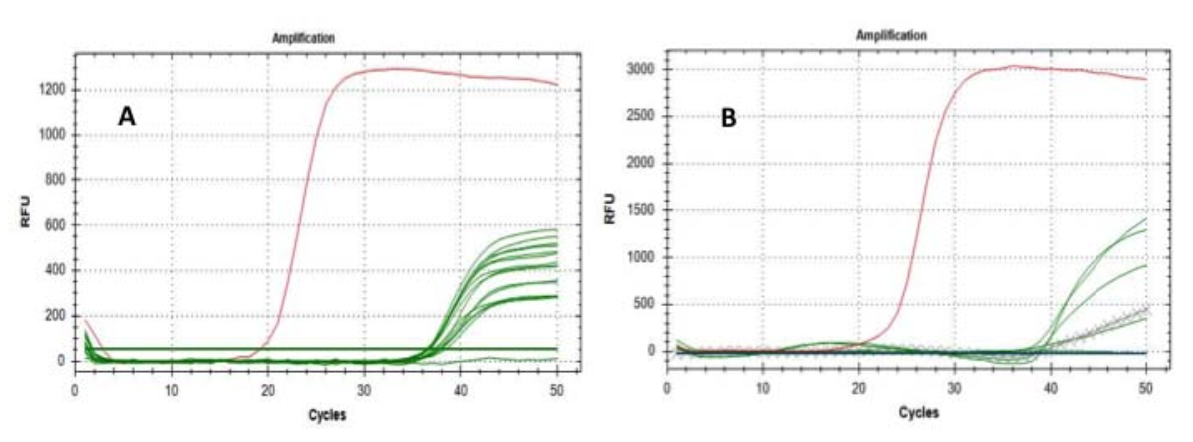
Fig. 2. Resultados positivos (marcados con X) para los genes E, N y RdRP obtenidos para la muestra de 34 PM10.Filtros probados en los segundos análisis paralelos.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Departamento de Química Industrial, Universidad de Bolonia, Viale del Risorgimento – 4, I-40136, Bolonia, Italia
e-mail: leonardo.setti@unibo.it
2. Centro Interdepartamental de Investigación Industrial “Fuentes Renovables, Medio Ambiente, Crecimiento Azul, Energía”,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Departamento de Biología, Universidad “Aldo Moro” de Bari, Bari, Italia
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Departamento de Ciencias Químicas y Farmacéuticas, Universidad de Trieste, Trieste, Italia
e-mail: barbierp@units.it
5. División de Investigación Ambiental, TCR TECORA, Milán, Italia
e-mail: mariagrazia.perrone@tcrtecora.com
6. Departamento de Ciencias de la Vida – Universidad de Trieste, Trieste, Italia
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. División de Medicina de Laboratorio, Hospital Universitario Giuliano Isontina (ASU GI), Trieste, Italia
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Sociedad Italiana de Medicina Ambiental (SIMA), Milán, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Departamento de Ciencias y Políticas Ambientales, Universidad de Milán, Milán, Italia
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Autor correspondiente:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Referencias
1. Organización Mundial de la Salud, Modos de transmisión del virus causante de la COVID-19: implicaciones para las recomendaciones de precaución de PCI, Informe científico; disponible en: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 de marzo de 2020)
2. Ministerio de Salud italiano, boletín diario sobre el brote de Covid-19 en Italia, disponible en http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. AEMA, Agencia Europea de Medio Ambiente, Informe sobre la calidad del aire en Europa 2019; n.º 10/2019; Agencia Europea de Medio Ambiente: Copenhague, Dinamarca, disponible en: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Exposición a la contaminación del aire y mortalidad por COVID-19 en Estados Unidos, disponible en: https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Sociedad Italiana de Medicina Ambiental (SIMA), Documento de posición sobre partículas y COVID-19,
Disponible en: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Is There a Plausible Role for Particulate Matter in the spreading of COVID-19 in Northern Italy?, BMJ Rapid Responses, 8 de abril de 2020, disponible en: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Generación de partículas finas fecales (PM2.5) contaminadas con el virus de la influenza aviar (VIA): detección del genoma y la infectividad, y cálculo de la inmisión. Microbiología Veterinaria. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La transmisión aérea podría haber influido en la propagación de los brotes de influenza aviar altamente patógena de 2015 en Estados Unidos. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Evaluación del impacto de los eventos de polvo en la incidencia del sarampión en el oeste de China. Atmospheric Environment. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI. Un modelo integrado para predecir la propagación atmosférica del virus de la fiebre aftosa. Epidemiol. Infect., 124, 577–590 (2000).
11. Glostera, J., Alexandersen, S. Nuevas direcciones: Transmisión aérea del virus de la fiebre aftosa en el entorno atmosférico, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA. Tasas de deposición de virus y bacterias por encima de la capa límite atmosférica. The ISME Journal. 12, 1154-1162 (2018).
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF. Estudio longitudinal del microbioma asociado con material particulado en una megaciudad. Genome Biology. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. La transmisión aérea puede tener
Desempeñó un papel en la propagación de los brotes de influenza aviar altamente patógena de 2015 en los Estados Unidos.
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Evaluación del impacto de los eventos de polvo en la incidencia del sarampión en el oeste de China. Atmospheric Environment. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF. Extracción optimizada de ADN y secuenciación metagenómica de comunidades microbianas aerotransportadas. Nat. Protoc. 10, 768-779 (2015).
17. Pan, M., Lednicky, JA, Wu, C.-Y., Recolección, dimensionamiento de partículas y detección de virus aerotransportados. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, descripción del producto, disponible en: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, descripción del producto, disponible en: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK y Mulders, DG (2020).
Detección del nuevo coronavirus 2019 (2019-nCoV) mediante RT-PCR en tiempo real. Eurosurveillance, 25(3), disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Original: https://doi.org/10.1101/2020.04.15.20065995
Hora de publicación: 18 de abril de 2020








