A síndrome respiratória aguda grave conhecida como doença COVID-19 – devido ao vírus SARS-CoV-2 – é reconhecida por se espalhar por meio de gotículas respiratórias e contatos próximos.[1] A carga da COVID-19 foi extremamente grave na Lombardia e no Vale do Pó (norte da Itália),[2] uma área caracterizada por altas concentrações de material particulado, que já são conhecidas por produzir efeitos negativos na saúde humana.[3] Os números regionais disponíveis para a Itália na data de 12 de abril mostram que cerca de 30% das pessoas atualmente positivas ainda vivem na Lombardia (cerca de 40% se considerarmos o total de casos confirmados desde o início da epidemia), seguidas pela Emília-Romanha (13,5%), Piemonte (10,5%) e Vêneto (10%).[2] Essas quatro regiões do Vale do Pó são responsáveis por 80% do total de mortes registradas na Itália e 65% das admissões em Unidades de Terapia Intensiva.[2]
Uma pesquisa realizada pela Escola de Saúde Pública de Harvard parece confirmar uma associação entre aumentos nas concentrações de PM e taxas de mortalidade devido à COVID-19 nos EUA[4] Em comunicações anteriores, levantamos a hipótese da possibilidade de o vírus SARS-CoV-2 poder estar presente em material particulado (PM) durante a propagação da infecção,[5,6] consistentemente com as evidências já existentes
disponível para outros vírus.[7-15] No entanto, a questão do microbioma associado às PM transportadas pelo ar, especialmente em ambientes urbanos, continua em grande parte pouco investigada,[16] e – no momento – ninguém ainda realizou estudos experimentais especificamente destinados a confirmar ou excluir a presença do SARS-CoV-2 nas PM.
Aqui, apresentamos os primeiros resultados das análises que realizamos em 34 amostras de PM10 externas/aéreas de um local industrial da província de Bérgamo, coletadas com dois amostradores de ar diferentes durante um período contínuo de 3 semanas, de 21 de fevereiro a 13 de março.
Seguindo a metodologia descrita por Pan et al. em 2019 (para coleta, dimensionamento de partículas e detecção de vírus aerotransportados),[17] amostras de MP foram coletadas em filtros de fibra de quartzo utilizando um amostrador de ar gravimétrico de baixo volume (38,3 l/min por 23 h), em conformidade com o método de referência EN12341:2014 para monitoramento de PM10. O material particulado foi retido em filtros com 99,9% de concentração típica.retenção de aerossol, devidamente armazenada e entregue ao laboratório de Genômica Aplicada e Comparativa da Universidade de Trieste. Dada a natureza “ambiental” da amostra, presumivelmente rica em inibidores de DNA polimerases, procedemos à extração de RNA utilizando o kit Quick RNA para microrganismos do solo fecal, adaptado ao tipo de filtro.[18] Metade do filtro foi enrolada, com a parte superior voltada para dentro.em um tubo de polipropileno de 5 ml, juntamente com as esferas fornecidas no kit. A partir do 1 ml inicial de tampão de lise, conseguimos obter cerca de 400 ul de solução, que foi então processada conforme definido pelos protocolos padrão, resultando em um eluato final de 15 ul. Posteriormente, 5 ul foram usados para o teste de SARS-CoV-2. Dada a origem particular da amostra, foi usado o qScript XLT 1-Step RT-qPCR ToughMix.[19] Os sistemas de amplificação foram os do protocolo desenvolvido por Corman et al, publicado no site da OMS [20].
O teste teve como objetivo explícito confirmar ou excluir a presença do RNA do SARS-CoV-2 em partículas. A primeira análise utilizou o "gene E" como marcador molecular e produziu um resultado positivo impressionante em 15 dos 16 filtros, mesmo que, como esperado, o Ct estivesse entre 36 e 38 ciclos.
Após isso, replicamos a análise em 6 dos filtros positivos (já positivos para o “gene E”) usando o “gene RtDR” como marcador molecular – que é altamente específico para SARS-CoV-2 – alcançando 5 resultados significativos de positividade; testes de controle para excluir falsa positividade também foram realizados com sucesso (Fig. 1).
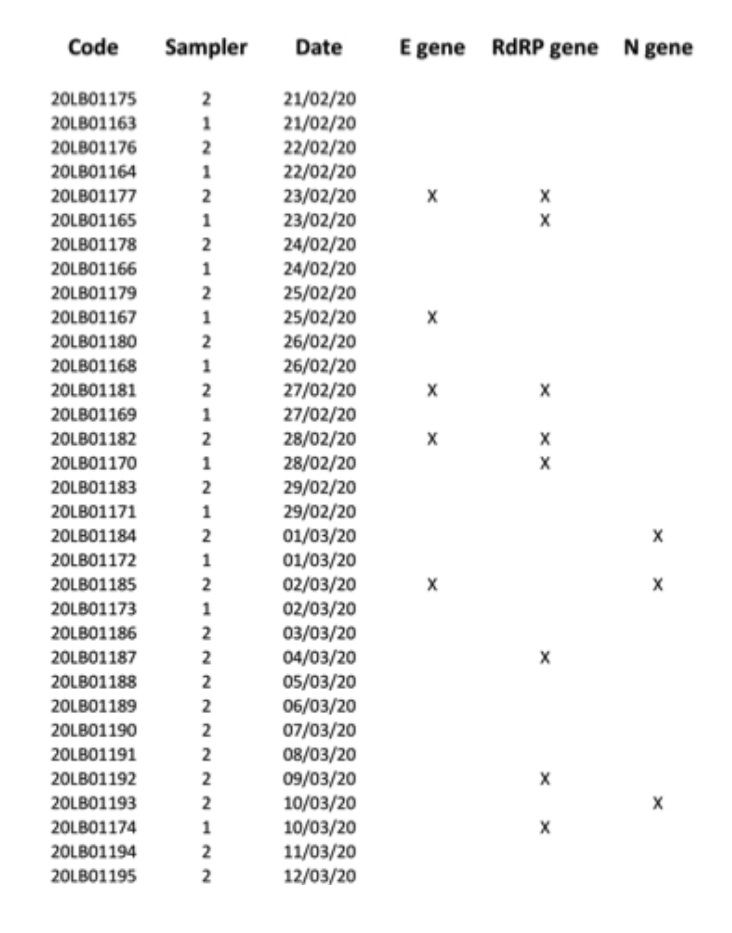
Para evitar o esgotamento do escasso material de amostragem disponível, os RNAs extraídos restantes foram entregues ao Hospital Universitário local (um dos centros clínicos autorizados pelo Governo Italiano para testes de diagnóstico de SARS-CoV-2), a fim de realizar um segundo teste cego paralelo. Este segundo laboratório clínico testou 34 extrações de RNA para os genes E, N e RdRP, relatando 7 resultados positivos para pelo menos um dos três genes marcadores, com positividade confirmada separadamente para todos os três marcadores (Fig. 2). Devido à natureza da amostra, e considerando que a amostragem não foi realizada para fins de diagnóstico clínico, mas para testes de poluição ambiental (levando também em consideração que os filtros foram armazenados por pelo menos quatro semanas antes de serem submetidos a análises genéticas moleculares, como(consequência do lockdown italiano), podemos confirmar que demonstramos razoavelmente a presença do RNA viral do SARS-CoV-2, detectando o "gene RtDR" altamente específico em 8 filtros. No entanto, devido à falta de materiais adicionais dos filtros, não conseguimos repetir um número suficiente de testes para demonstrar positividade para todos os 3 marcadores moleculares simultaneamente.
Esta é a primeira evidência preliminar de que o RNA do SARS-CoV-2 pode estar presente em partículas externas, sugerindo que, em condições de estabilidade atmosférica e altas concentrações de MP, o SARS-CoV-2 poderia criar aglomerados com MP externas e – ao reduzir seu coeficiente de difusão – aumentar a persistência do vírus na atmosfera. Novas confirmações desta preliminarAs evidências estão em andamento e devem incluir avaliações em tempo real sobre a vitalidade do SARS-CoV-2, bem como sua virulência quando adsorvido em material particulado. No momento, não é possível fazer suposições sobre a correlação entre a presença do vírus em partículas e a progressão do surto de COVID-19. Outras questões a serem abordadas especificamente são as concentrações médias de partículas eventualmente presentes.necessário para um potencial “efeito de reforço” do contágio (caso seja confirmado que o PM pode atuar como um “transportador” para os núcleos de gotículas virais), ou mesmo a possibilidade teórica de imunização consequente a exposições a doses mínimas em limiares mais baixos de PM.
Fig.1 Curvas de amplificação dos genes E (A) e RdRP (B): as linhas verdes representam os filtros testados; as linhas cruzadasrepresenta extrações do filtro de referência; as linhas vermelhas representam a amplificação das amostras positivas.
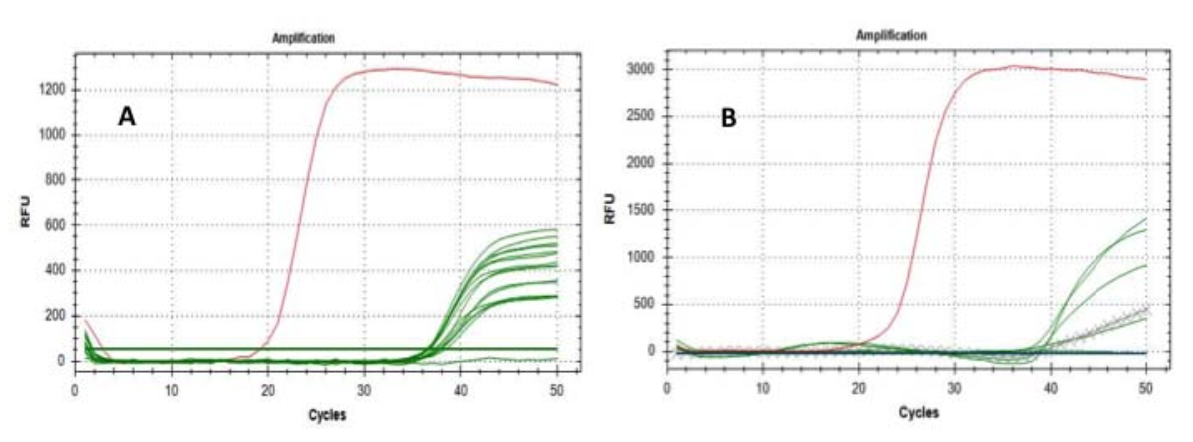
Fig.2. Resultados positivos (marcados com X) para os genes E, N e RdRP obtidos para todas as 34 amostras de PM10filtros testados nas segundas análises paralelas.
 Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
Leonardo Setti1, Fabrizio Passarini2, Gianluigi De Gennaro3, Pierluigi Barbieri4, Maria Grazia Perrone5, Massimo Borelli6, Jolanda Palmisani3, Alessia Di Gilio3, Valentina Torboli6, Alberto Pallavicini6, Maurizio Ruscio7, Prisco Piscitelli8, Alessandro Miani8,9
1. Departamento de Química Industrial, Universidade de Bolonha, Viale del Risorgimento – 4, I-40136, Bolonha, Itália
e-mail: leonardo.setti@unibo.it
2. Centro Interdepartamental de Investigação Industrial “Fontes Renováveis, Ambiente, Crescimento Azul, Energia”,
University of Bologna, Rimini, Italy e-mail: fabrizio.passarini@unibo.it
3. Departamento de Biologia, Universidade “Aldo Moro” de Bari, Bari, Itália
e-mail: gianluigi.degennaro@uniba.it; alessia.digilio@uniba.it; jolanda.palmisani@uniba.it
4. Departamento de Ciências Químicas e Farmacêuticas, Universidade de Trieste, Trieste, Itália
e-mail: barbierp@units.it
5. Divisão de Pesquisa Ambiental, TCR TECORA, Milão, Itália
e-mail: mariagrazia.perrone@tcrtecora.com
6. Departamento de Ciências da Vida – Universidade de Trieste, Trieste, Itália
e-mail: borelli@units.it; torboli@units.it; pallavic@units.it
7. Divisão de Medicina Laboratorial, Hospital Universitário Giuliano Isontina (ASU GI), Trieste, Itália
email: maurizio.ruscio@asugi.sanita.fvg.it
8. Sociedade Italiana de Medicina Ambiental (SIMA), Milão, Itália
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
9. Departamento de Ciência e Política Ambiental, Universidade de Milão, Milão, Itália
e-mail: priscofreedom@hotmail.com; alessandro.miani@unimi.it
Autor correspondente:
Leonardo Setti, Department of Industrial Chemistry, University of Bologna Viale del Risorgimento 4, 40136, Bologna, Italy; e-mail: leonardo.setti@unibo.it
Referências
1. Organização Mundial da Saúde, Modos de transmissão do vírus causador da COVID-19: implicações para as recomendações de precaução de PCI, Resumo científico; disponível em: https://www.who.int/newsroom/commentaries/detail/modes-of-transmission-of-virus-causing-covid-19-implications-for-ipcprecaution-recommendations (29 de março de 2020)
2. Ministério da Saúde italiano, boletim diário sobre o surto de Covid-19 na Itália, disponível em http://www.salute.gov.it/imgs/C_17_notizie_4451_0_file.pdf
3. AEA, Agência Europeia do Ambiente, Relatório sobre a Qualidade do Ar na Europa 2019; n.º 10/2019; Agência Europeia do Ambiente: Copenhaga, Dinamarca, disponível em: https://www.eea.europa.eu/publications/airquality-in-europe-2019
4. Xiao Wu, Rachel C. Nethery, M. Benjamin Sabath, Danielle Braun, Francesca Dominici, Exposição à poluição do ar e mortalidade por COVID-19 nos Estados Unidos, disponível em: https://projects.iq.harvard.edu/files/covid-pm/files/pm_and_covid_mortality.pdf
5. Sociedade Italiana de Medicina Ambiental (SIMA), Documento de Posição sobre Matéria Particulada e COVID-19,
disponível em: http://www.simaonlus.it/wpsima/wp-content/uploads/2020/03/COVID_19_positionpaper_ENG.pdf
6. Setti L., Passarini F., De Gennaro G., Barbieri P., Perrone MG, Piazzalunga A., Borelli M., Palmisani J., Di Gilio A, Piscitelli P, Miani A., Existe um papel plausível para material particulado na disseminação de COVID-19 no norte da Itália?, BMJ Rapid Responses, 8 de abril de 2020, disponível em: https://www.bmj.com/content/368/bmj.m1103/rapid-responses
7. Sedlmaier, N., Hoppenheidt, K., Krist, H., Lehmann, S., Lang, H., Buttner, M. Geração de material particulado fino fecal (PM2,5) contaminado pelo vírus da influenza aviária (VIA): detecção de genoma e infectividade e cálculo de imissão. Microbiologia Veterinária. 139, 156-164 (2009)
8. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. A transmissão aérea pode ter desempenhado um papel na disseminação dos surtos de gripe aviária altamente patogênica de 2015 nos Estados Unidos. Sci Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
9. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Avaliação do impacto de eventos de poeira na incidência de sarampo no oeste da China. Ambiente Atmosférico. 157, 1-9 (2017)
10. Sorensen, JH, Mackay, DKJ, Jensen, C. Ø., Donaldson, AI Um modelo integrado para prever a propagação atmosférica do vírus da febre aftosa Epidemiol. Infect., 124, 577–590 (2000)
11. Glostera, J., Alexandersen, S. Novas Direções: Transmissão Aérea do Vírus da Febre Aftosa Ambiente Atmosférico, 38 (3), 503-505 (2004)
12. Reche, I., D'Orta, G., Mladenov, N., Winget, DM, Suttle, CA Taxas de deposição de vírus e bactérias acima da camada limite atmosférica. The ISME Journal. 12, 1154-1162 (2018)
13. Qin, N., Liang, P., Wu, C., Wang, G., Xu, Q., Xiong, X., Wang, T., Zolfo, M., Segata, N., Qin, H., Knight, R., Gilbert, JA, Zhu, TF. Levantamento longitudinal do microbioma associado a material particulado em uma megacidade. Biologia do Genoma. 21, 55 (2020)
14. Zhao, Y., Richardson, B., Takle, E., Chai, L., Schmitt, D., Win, H. A transmissão aérea pode ter
desempenhou um papel na disseminação dos surtos de gripe aviária altamente patogênica de 2015 nos Estados Unidos.
Rep. 9, 11755. https://doi.org/10.1038/s41598-019-47788-z (2019)
15. Ma, Y., Zhou, J., Yang, S., Zhao, Y., Zheng, X. Avaliação do impacto de eventos de poeira na incidência de sarampo no oeste da China. Ambiente Atmosférico. 157, 1-9 (2017)
16. Jiang, W., Laing, P., Wang, B., Fang, J., Lang, J., Tian, G., Jiang, J., Zhu, TF Extração otimizada de DNA e sequenciamento metagenômico de comunidades microbianas aéreas. Nat. Protoc. 10, 768-779 (2015)
17. Pan, M., Lednicky, JA, Wu, C.-Y., Coleta, dimensionamento de partículas e detecção de vírus transportados pelo ar. Journal of Applied Microbiology, 127, 1596-1611 (2019)
18. Zymoresearch Ldt, descrição do produto, disponível em: https://www.zymoresearch.com/products/quick-rnafecal-soil-microbe microprep-kit
19. Quantabio Ltd, descrição do produto, disponível em: https://www.quantabio.com/qscript-xlt-1-steprt-qpcr-toughmix
20. Corman, VM, Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, DK, & Mulders, DG (2020).
Detecção do novo coronavírus de 2019 (2019-nCoV) por RT-PCR em tempo real. Eurosurveillance, 25(3), disponível em: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
Original: https://doi.org/10.1101/2020.04.15.20065995
Data de publicação: 18/04/2020








